Les équipes de Google DeepMind ont annoncé récemment la sortie d’un jeu de données d’observation de la Terre novateur, issu de l’assimilation d’une variété de données satellitaires de différents capteurs au sein d’un modèle de fondation. Sans entrer dans les détails (un post sur Medium1 et la documentation technique2 le font très bien), ce jeu de données, baptisé « Satellite Embedding », compresse l’information spectrale et radar d’une année entière d’observations en un vecteur à 64 dimensions, livré à 10 mètres de résolution. Autrement dit : chaque pixel de 10 × 10 m contient 64 valeurs qui résument tout ce qui a été observé par les satellites sur une année donnée.

Ce qui est rigolo, c’est qu’on n’a aucun a priori sur ce que peut représenter chacune de ces dimensions dans le monde physique, contrairement, par exemple, à la bande 2 de Sentinel-2, d’une longueur d’onde de 490 nm et liée au spectre d’absorption de la chlorophylle, ou à la rétrodiffusion radar de Sentinel-1, sensible à l’angle de la pente des reliefs. Pire encore, on ne sait pas comment l’information temporelle est encodée : est-ce que la date d’une augmentation du NDVI, que l’on sait reliée à la croissance d’un couvert végétal, sera portée par une ou plusieurs composantes du vecteur ?
Le jeu satellite embeddings agrège donc une année d’observations multi-capteurs (optique + radar) en vecteurs latents ; on n’interprète pas chaque dimension physiquement, mais ces représentations condensent des motifs multi-sources et multi-temporels du couvert observé (structure, textures, phénologie, mosaïques d’occupation des sols) à la résolution des données d’entrée. Elles sont ainsi de bons proxies de l’état actuel du paysage, sans garantir qu’une dimension unique corresponde à un processus écologique précis.
Dans tous les cas, on sait que ce jeu de données contient une quantité phénoménale d’information, à une résolution très intéressante pour bon nombre d’applications.
Pourquoi cette note ?
Une des applications que j’aimerais explorer, c’est la cartographie d’espèces végétales. Et dans ce post, c’est justement une exploration rapide de cette idée que je présente.
Assez classiquement, la cartographie d’espèces passe par des approches de modélisation. Pourquoi modéliser ? Parce qu’il est difficile d’échantillonner l’espace assez finement pour dessiner des cartes « à la main ». On s’appuie donc sur des variables environnementales, généralement bien décrites et disponibles à bonne résolution spatiale et temporelle, pour estimer la probabilité de présence d’une espèce donnée. On peut pour cela mobiliser les paramètres écologiques/édaphiques de l’espèce (par ex. intervalles de température, pluviométrie annuelle, types de sols préférentiels, pente, altitude, etc.) afin d’identifier où l’espèce peut survivre et prospérer. C’est l’approche utilisée notamment par EcoCrop (FAO)3, centré en partie sur des espèces d’intérêt agronomique.
Quand ces paramètres sont mal connus ou incomplets, on peut adopter une approche fondée sur les données : on utilise des relevés botaniques qui indiquent la présence (l’absence étant, par définition, plus délicate à établir) à un endroit donné, on associe ces positions aux covariables au même endroit, puis on calibre des modèles de distribution d’espèces (Species Distribution Models, SDMs) pour extrapoler la probabilité de présence ailleurs. Il existe plusieurs modèles ; l’un des plus populaires est MaxEnt (pour « Maximum Entropy », le principe mathématique sur lequel il repose).
Pourquoi ce détour ? Parce que si l’on peut utiliser des variables environnementales spatialisées dans des SDMs pour estimer la répartition d’une espèce, il n’y a aucune raison de ne pas essayer un produit comme les Satellite Embeddings, qui résument une quantité considérable d’information observable depuis l’espace.
Distinction importante : alors que l’utilisation de variables environnementales permet surtout de définir où l’espèce pourrait se trouver (aire de répartition potentielle), les satellite embeddings pourraient, en principe, aider à répondre à où l’espèce se trouve aujourd’hui, car ils proviennent en grande partie d’observations directes du couvert végétal (signal « actuel »).
Je présente donc ci-après quelques essais rapides utilisant les satellite embeddings comme covariables dans un SDM MaxEnt, pour prédire la probabilité d’occurrence de quelques espèces végétales en Martinique.
Données et méthodes
Je commence par récupérer les satellite embeddings. Pour défricher rapidement, j’en fais l’extraction en GeoTIFF depuis Google Earth Engine, à une résolution dégradée à 100 m (agrégation par moyenne).

Trois espèces m’intéressent ici : l’arbre à pain (Artocarpus altilis) et le cocotier (Cocos nucifera) — parce que différences d’étagements, parce que des collègues s’y intéressent, et parce qu’on dispose d’un nombre d’observations important (n=341 et n=181 respectivement) — ainsi que la fleur boule montagne (Lobelia conglobata), endémique stricte de la Martinique, pour laquelle les observations sont bien moindres (n=64) et sur un étagement bien plus élevé.
Pour entraîner un SDM MaxEnt4, il faut des présences. Je lance donc des requêtes sur GBIF5 et iNaturalist6. Face aux 64 dimensions du jeu Satellite Embeddings (satemb), ce volume paraît suffisant pour une première exploration en limitant le risque de mal-conditionnement.
Avec un peu de Python (et un soupçon de magie noire), je prépare les fichiers d’entrée du SDM : les variables satemb, mais aussi des variables environnementales (env) — Tmin/Tmax/Tmoy annuelles (interpolation Météo-France 2023), pluie annuelle (stations DIREN + Météo-France, 7), altitude, pente, aspect, rugosité dérivées du SRTM/Copernicus 30 m, sur même zone d’étude, même grille 100 m et masque terrestre pour tous les rasters. J’entraîne ensuite MaxEnt v3.4.4 sur trois jeux de covariables : satemb, env et satemb+env, avec une validation croisée en cinq plis ; les chiffres reportés sont des moyennes ± écart-types de ces 5 plis.
L’ensemble des scripts et données pour répliquer l’analyse (et en lancer d’autres) est mis à disposition sur Zenodo : 10.5281/zenodo.16994138.
Résultats
Pour l’arbre à pain (A. altilis, Fig. 3), le modèle fondé sur les satellite embeddings seuls obtient l’AUC la plus élevée (0,833 ± 0,029), devant le modèle env (0,661 ± 0,044), soit un écart absolu de +0,172. Le modèle mixte est à 0,803 ± 0,055, donc en dessous de satemb. À grille identique (100 m) pour tous les prédicteurs, ces résultats suggèrent que les indices satellitaires condensés par les embeddings discriminent mieux les présences du fond que les seules variables abiotiques utilisées embarquées dans le modèle env. L’ajout des variables environnementales n’apporte pas de gain mesurable dans ce cas (probable redondance d’information + complexité accrue).
Pour le cocotier (C. nucifera, Fig. 4), l’écart entre satemb et env est plus faible que chez l’arbre à pain : 0,833 ± 0,036 contre 0,767 ± 0,045, soit +0,066 d’AUC pour satemb. Le mixte atteint 0,809 ± 0,045, intermédiaire entre les deux. Ici, les variables abiotiques (p. ex. altitude, pluie) portent déjà une part substantielle du signal, et les embeddings ajoutent un surcroît d’information sans toutefois dépasser nettement la combinaison.
Pour L. conglobata (Fig. 5, espèce localisée en altitude en Martinique), les deux approches sont très performantes et la combinaison progresse encore : satemb 0,907 ± 0,015, env 0,906 ± 0,059, et mixte 0,933 ± 0,046 (soit +0,026 vs satemb). Lecture possible : l’enveloppe abiotique (altitude, exposition, humidité) cadre déjà bien la niche, et les embeddings ajoutent des indices d’occupation actuelle du paysage (structure de canopée, mosaïques agri-urbaines, phénologie), d’où un gain quand on les combine. A noter cependant un nombre d’observations retenues par le modèle (neff) particulièrement réduit, lié au regroupement des occurrences d’un même pixel, ce qui invite à considérer ces résultats avec prudence.



satemb, centre : modèle env, droite : modèle satemb+env
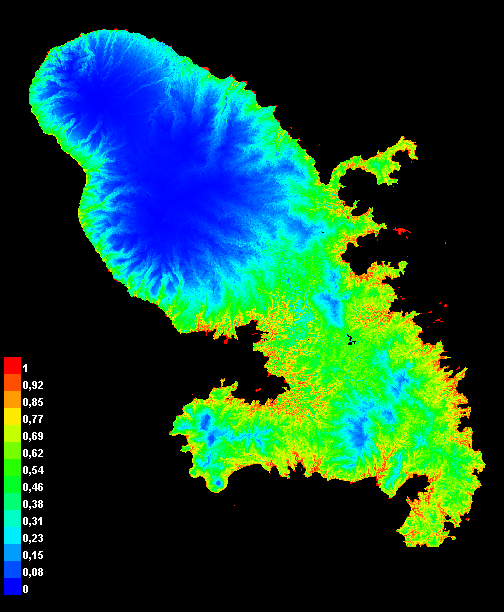

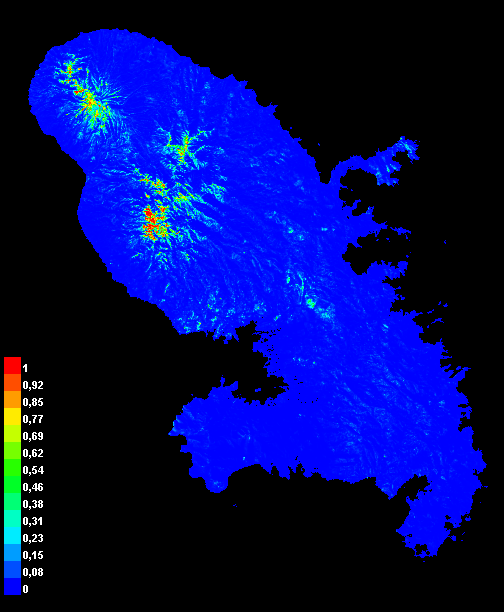
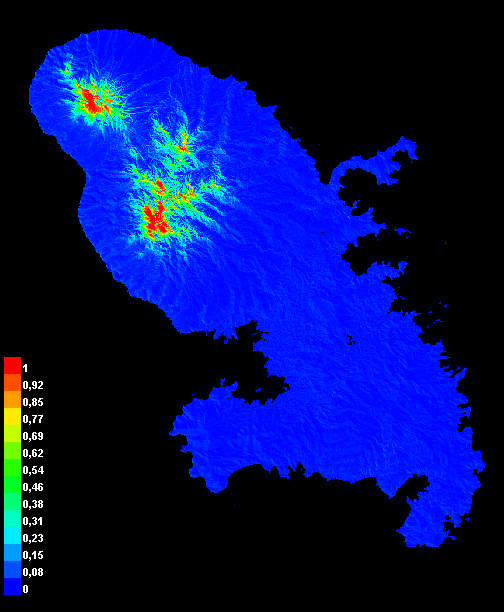
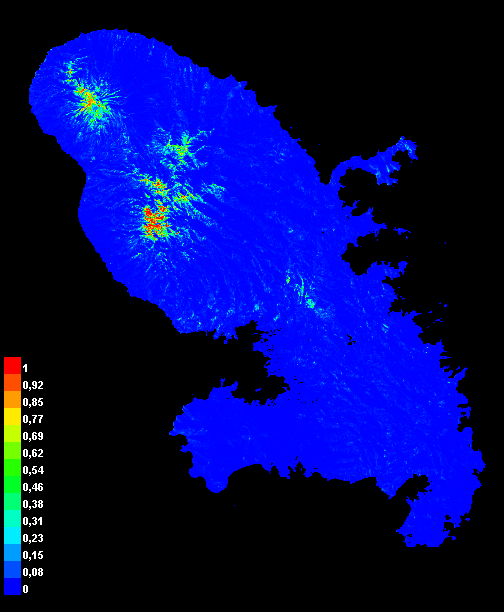
satemb | env | satemb+env | |
| A. altilis (neff = 168) | 0,833 (± 0,029) | 0,661 (± 0,044) | 0,803 (± 0,055) |
| C. nucifera (neff = 78) | 0,833 (± 0,036) | 0,767 (± 0,045) | 0,809(± 0,045) |
| L. conglobata (neff = 31) | 0,907 (± 0,015) | 0,906 (± 0,059) | 0,933 (± 0,046) |
En lecture conceptuelle, l’approche env définit une enveloppe abiotique (où l’espèce pourrait vivre), tandis que l’approche satemb fournit des indices d’occupation actuelle (où le paysage ressemble aujourd’hui aux sites observés), complémentarité visible en Fig. 3–4 ; le modèle mixte (env+satemb) approxime l’intersection de ces deux informations — conditions favorables ET indices compatibles — sans pour autant conclure à une « présence certaine ». Pour l’exploiter, on peut envisager de croiser les deux surfaces après choix d’un seuil validé en CV (p. ex. 10è centile des présences) et l’on cartographie trois classes : Potentiel ? Présent (cœur réalisé), Potentiel \ Présent (réservoir latent) et Présent \ Potentiel (hors enveloppe) ; l’intérêt est alors bien de lire les modèles ensemble plutôt que d’interpréter chaque carte isolément.
Limites et discussion
Cette approche exploratoire souffre néanmoins d’un certain nombre de limites :
- Échantillonnage des présences
- Echatillonage participatif et biais d’accessibilité (proximité routes/bourgs) non pondéré ; pas de bias file / target-group background utilisé.
- Pas de thinning spatial : malgré le “1 point par pixel” implicite de MaxEnt, des clusters peuvent subsister et peuvent biaiser l’ajustement.
- Covariables et échelle
- Embeddings : approche boîte noire ? interprétabilité limitée variable par variable ; forte colinéarité possible entre dimensions : feature engineering/PCA/UMAP pour tester si 10–15 composantes suffisent ?
- Variables
env: jeu réduit (pas de sols notamment) ; d’autres facteurs abiotiques/édaphiques non pris en compte. - Résolution unique 100 m choisie pour la praticité volume de données/temps de calcul : sensibilité à l’échelle (i.e. 30m natif SRTM vs 100m, etc.) non évaluée.
- Paramétrage et protocole d’entraînement
- Régularisation non optimisée (valeurs par défaut) ; pas de sélection parcimonieuse des prédicteurs. MaxEnt ? processus ponctuel de Poisson régularisé : régler la pénalité et les feature classes limite le sur-apprentissage, surtout avec 64 dims.
- Cohérence temporelle
- Présences multi-années non synchronisées avec les covariables : acceptable ici (espèces pluriannelles à pérennes), mais pour des annuelles il faudrait aligner l’année d’observation avec l’année des variables.
- Validité et portée des résultats
- CV spatiale en blocs (au lieu de k-fold aléatoire) pour limiter l’optimisme lié à l’autocorrélation et tester la transférabilité spatiale.
- L’AUC (définie ici via “predicted area” au sens MaxEnt) est utile mais peut être indulgente en présence d’autocorrélation spatiale et de prévalence très faible.
- Conclusions dépendantes de l’espèce, de la zone et de l’échelle ; transférabilité non testée.
Ces limites n’invalident pas fondamentalement les tendances observées, cependant, elles bornent la portée des résultats et indiquent les priorités d’amélioration pour une itération suivante.
Conclusion
À emprise et résolution communes (100 m), les satellite embeddings apportent un signal propre exploitable en SDM : ils suffisent à eux seuls pour distinguer les présences du fond sur deux espèces testées (A. altilis, C. nucifera), et, pour L. conglobata, la combinaison avec les variables abiotiques montre un léger mieux — à confirmer compte tenu du neff réduit et de la CV non spatiale. Le signal porté par ces représentations latentes — proxys d’observations multisources du couvert à 10 m — est donc complémentaire des gradients climato-topographiques : parfois suffisant seul, souvent utile en appui selon le profil écologique.
Méthodologiquement, ces résultats suggèrent un workflow pragmatique : (i) considérer les embeddings comme baseline forte, (ii) n’ajouter que quelques variables abiotiques non redondantes quand l’écologie l’exige, et (iii) fiabiliser par CV spatiale, régularisation et correction du biais d’échantillonnage. Au-delà de l’AUC, des indicateurs comme le Continuous Boyce Index, le partial ROC et des cartes MESS aideront à juger robustesse, calibration et extrapolation.
Enfin, parce que les embeddings sont disponibles par année, ils ouvrent des perspectives pratiques (à tester) : suivi interannuel de l’aire effectivement occupée, détection de déplacements (expansion/contraction), mise en évidence de foyers « hors enveloppe » (plantations, micro-refuges) et priorisation de prospections (zones « potentiel haut ? présent haut »). Cela plaide pour des SDM opérationnels où les embeddings jouent un rôle central, en propre ou en complément de l’abiotique.
Références
- Google Earth Team (2025) « AI-powered pixels: Introducing Google’s Satellite Embedding dataset » https://medium.com/google-earth/ai-powered-pixels-introducing-googles-satellite-embedding-dataset-31744c1f4650 ↩︎
- Google Deep Mind [internet] Satellite Embedding V1 https://developers.google.com/earth-engine/datasets/catalog/GOOGLE_SATELLITE_EMBEDDING_V1_ANNUAL?hl=fr. Accessed on 2025-8-29. ↩︎
- FAO. [Internet] « ECOCROP Database of Crop Constraints and Characteristics » https://gaez.fao.org/pages/ecocrop. Accessed on 2025-8-29. ↩︎
- Steven J. Phillips, Miroslav Dudík, Robert E. Schapire. [Internet] Maxent software for modeling species niches and distributions (Version 3.4.1). Available from url: http://biodiversityinformatics.amnh.org/open_source/maxent/. Accessed on 2025-8-29. ↩︎
- https://www.gbif.org ↩︎
- iNaturalist. Available from https://www.inaturalist.org. Accessed 2025-8-29 ↩︎
- Lavarenne, Jérémy, 2024, « Martinique daily interpolated rainfall, 1980-2021, obtained via ordinary kriging », https://doi.org/10.18167/DVN1/KJEA4Q, CIRAD Dataverse, V1 ↩︎